ATAC-Seq
背景
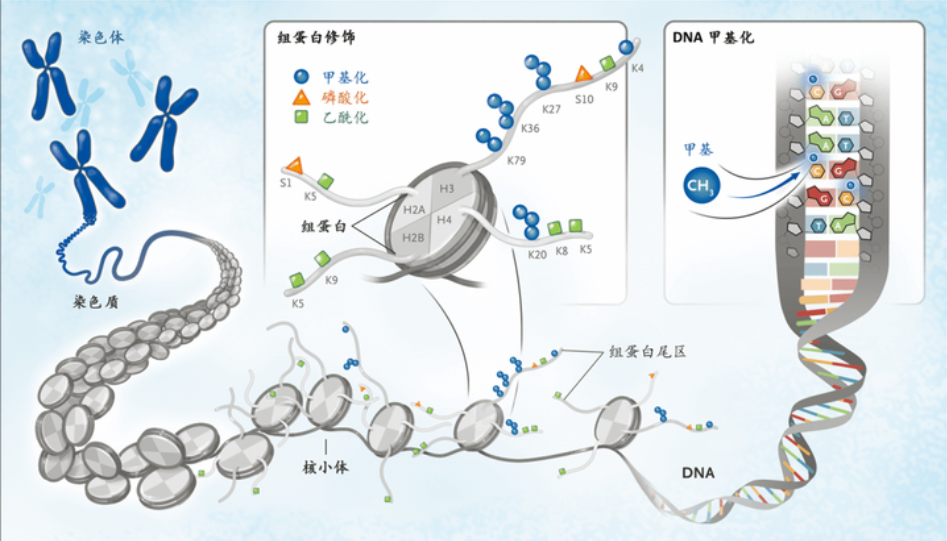
- DNA一圈圈绕在八个组蛋白上,形成核小体。
- 一个个核小体构成串珠样结构,进一步折叠聚合,形成染色体。
- 基因的转录需要将DNA的高级结构打开一部分,即表达基因的区域解开,而这一过程,主要由染色体组蛋白的修饰(尤其是乙酰化)来实现的,这部分打开的染色质就是开放染色质。
- 染色质一旦打开,一些调控蛋白,如转录因子与之结合。染色质的这种特性就是染色质的可及性
- 故染色质的可及性反应的就是调控因子与开放染色质结合的状态,与转录调控密切相关
如何找开放的染色质区域
转座酶Tn5
- DNA转座是一种由DNA转座酶介导,把DNA序列从染色体的一个区域插入到另外一个区域的现象。这个过程需要插入的位点的染色质是开放的。
将NGS接头连接到转座酶,携带这些接头的转座酶进入细胞核后,在染色质开放区域自由发挥,使染色质断裂并将这些接头插入到开放的染色质区域中,后利用已知序列的标签进行PCR后测序,就知道哪些区域是开放染色质了。
ATAC-seq和Chip-seq的区别
- ChIP-seq是实验前明确有一个感兴趣的转录因子,根据目标转录因子设计抗体,验证感兴趣的转录因子是否与DNA存在相互作用
- ATAC-seq没有具体到某个转录因子,是在全基因组范围内检测染色质的开放程度,可以得到全基因组范围内的蛋白质可能结合的位点信息,用这个技术方法与其他方法结合是想去筛感兴趣的调控因子。
常见的染色质开放区
常见的染色质开放区域主要是基因上游的启动子和远端的调控元件如增强子和沉默子
启动子是靠近转录起始点(TSS)的DNA区域,它包含转录因子的结合位点(TFBS),故转录因子能够结合在启动子上TFBS,招募RNA聚合酶进而转录基因
增强子一般位于启动子下游或上游1Mb的DNA区域,转录因子与增强子结合,并与启动子区域接触时,能够促进基因的转录。相反,沉默子会减少或抑制基因的表达
ATAC-seq可以帮助识别启动子区域、潜在的增强子或沉默子,即ATAC-seq中的peak,往往是启动子、增强子序列,以及一些反式调控因子结合的位点。
应用
鉴定重要的转录因子
- ATAC捕获的染色质开放区域一般是正在转录的那部分DNA序列的上下游,得到这些序列就可以对富集到的序列结合motif分析,识别哪种转录因子参与了基因表达调控。
- motif分析用于识别DNA、RNA或蛋白质序列中的特定、重复的模式,这些模式通常与生物学上重要的结构、功能或调控元件相关联。这些模式通常称为”motif”或”序列模式”,它们可以是短的核苷酸序列(例如DNA序列中的TF结合位点或蛋白质序列中的结构域),也可以是更复杂的结构模式(如RNA二级结构中的stem-loop或蛋白质三维结构中的保守的结构域)
- ATAC捕获的染色质开放区域一般是正在转录的那部分DNA序列的上下游,得到这些序列就可以对富集到的序列结合motif分析,识别哪种转录因子参与了基因表达调控。
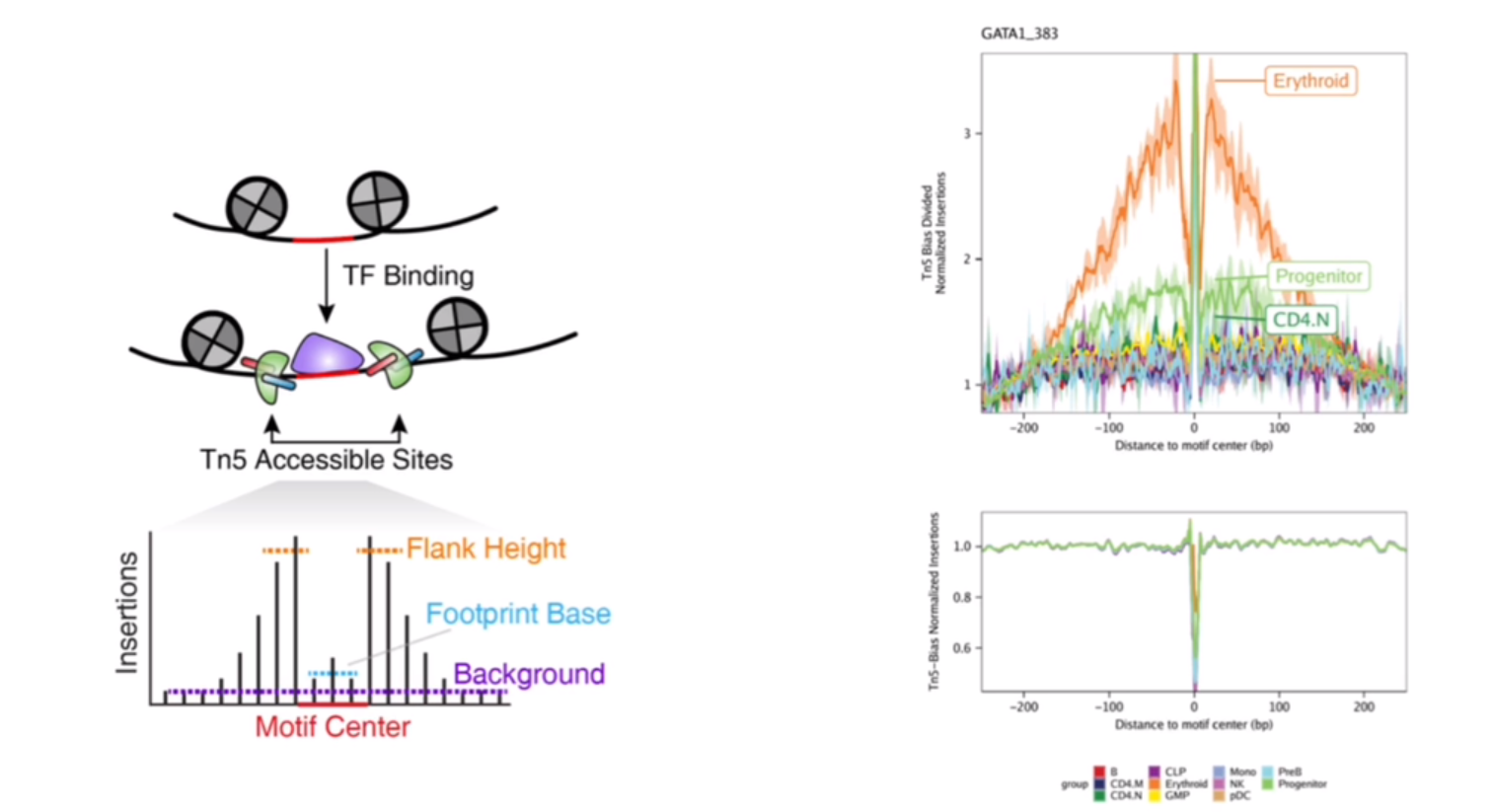
生成转录因子结合区域的特征(footprinting)
- 转录因子结合在DNA上后,它占有的空间阻碍了转座酶Tn5酶切在其他无核小体区域,这样就会留下一个一个小区域,称为足迹(footprint),在这些区域中,reads由高覆盖率峰值突然下降,如图,在转录因子结合的位置是一个“低谷”的形状。
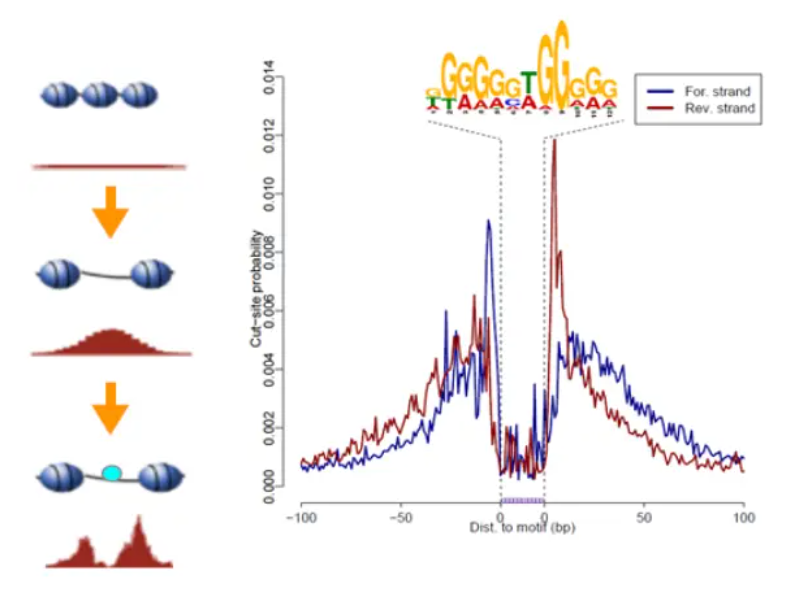
得到核小体位置
分析
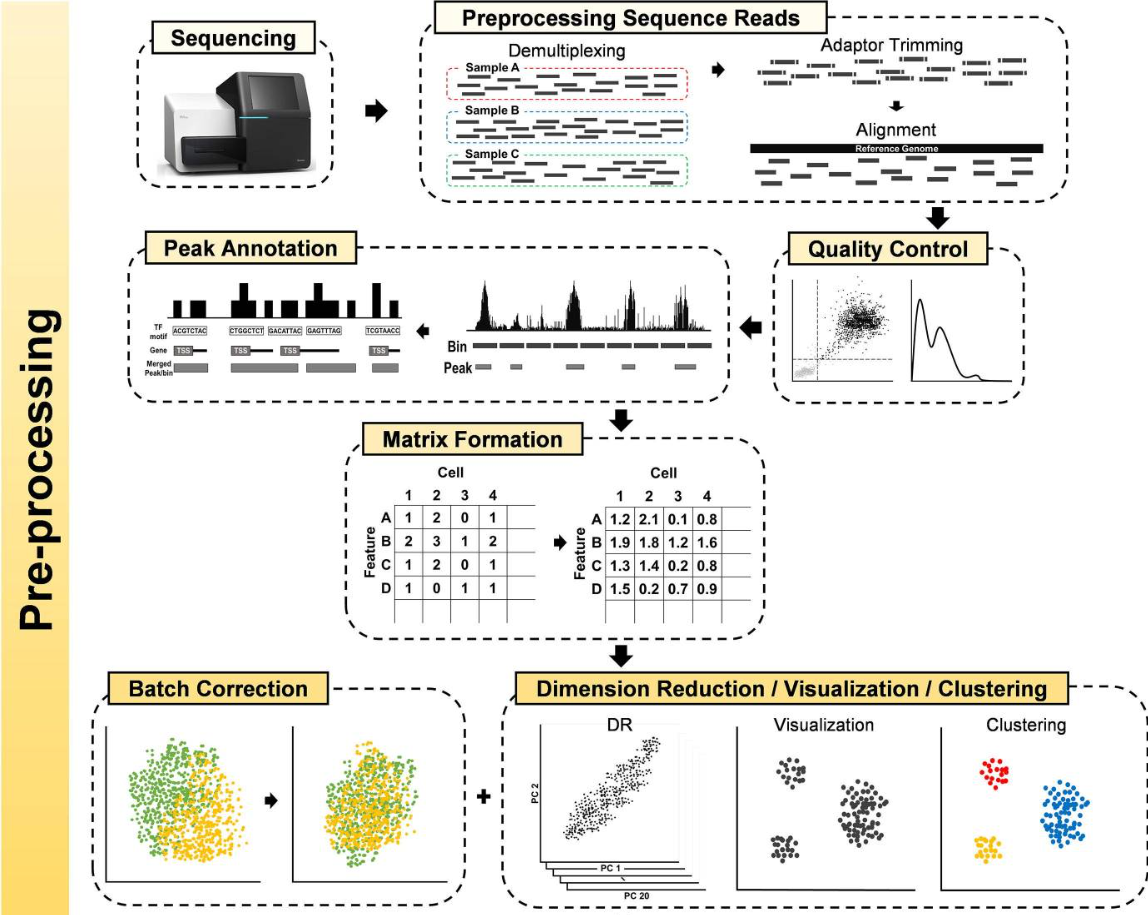
Preprocessing Sequence Reads
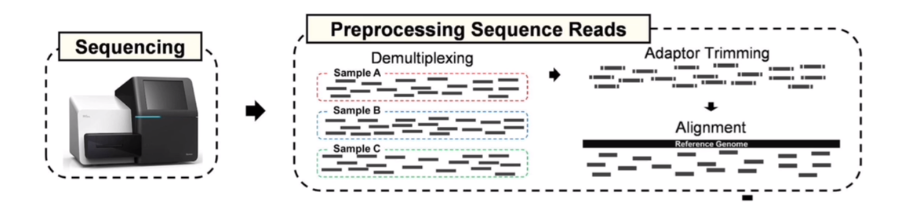
- Demultiplexing:将每个样本的reads区分开
- Adaptor Trimming:去除测序接头
- Alignment:将得到reads比对到基因组上
QC
- Filter Cells
- Filter fragments
Batch correction
- Data integration(LIGER、Harmony)
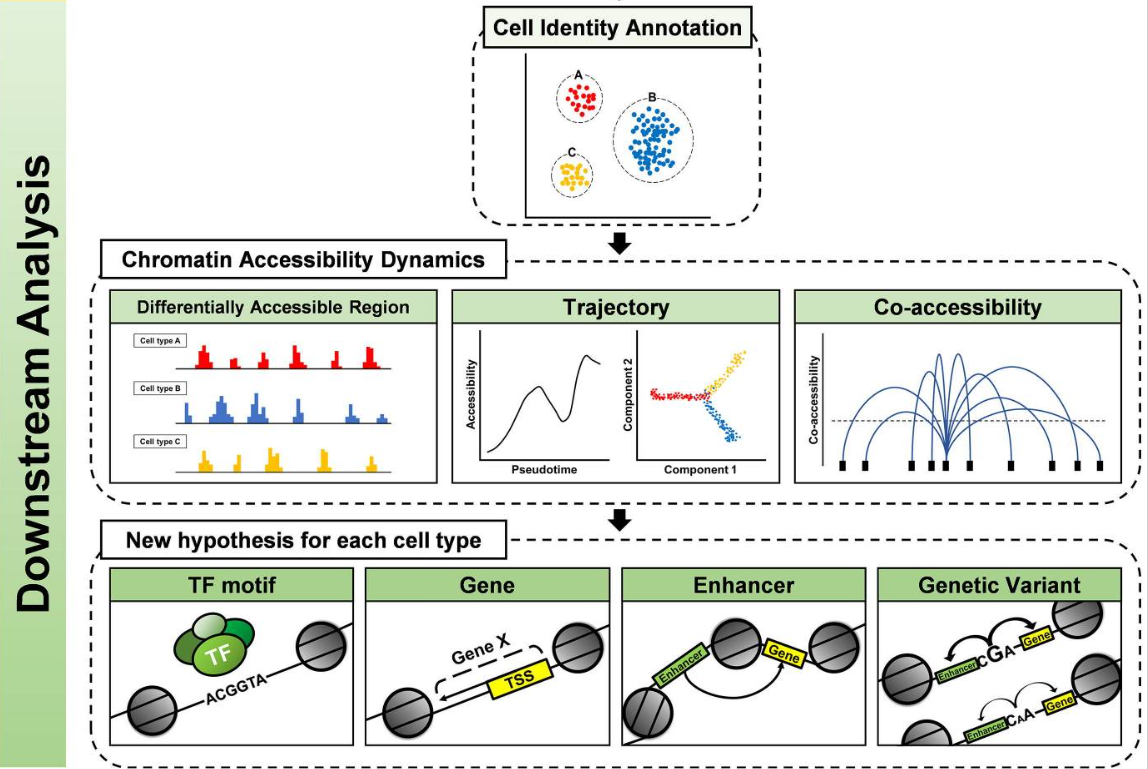
Cell type annotation
- 由于ATAC-seq的特征中,不知道其基因是做什么的,需要进行特征转换
- 将基因组上的peak转换成类似基因活性概念的特征
Differential analysis
- 得到差异基因组的region,但不知道其功能
- GREAT tool:查看差异的peak
Trajectory Inference
- 推断发育轨迹
- 查看某个peak随着伪时间的推进的强弱
Co-accessibility
- 查看哪些peak有协同作用,这些peak在基因组上可能有互作作用(Cicero)
Motif analysis
- 查看motif在哪个细胞类型中是特异性富集的,从而推断转录因子(ChromVAR)
Footprint analysis
- 右图一种颜色代表一种细胞类型